Utvrđivanje da li će se i kako će se nanočestica i protein povezati jedni sa drugima, važan je korak ka mogućnosti dizajniranja antibiotika i antivirusnih lekova na zahtev, a kompjuterski model razvijen na Univerzitetu u Mičigenu to može da uradi.
Novi alat bi mogao pomoći u pronalaženju načina za zaustavljanje infekcija otpornih na antibiotike i novih virusa – i pomoći u dizajnu nanočestica za različite svrhe.
„Samo u 2019. broj ljudi koji su umrli od rezistencije na antimikrobne lekove bio je 4,95 miliona. I pre COVID-a, koji je pogoršao problem, studije su pokazale da će do 2050. broj umrlih od rezistencije na antibiotike biti 10 miliona“, rekla je Angela Violi, Arthur F. Thurnau, profesor mašinstva, i odgovarajući autor studije koja je napravila naslovnicu Nature Computational Science.
„U mom idealnom scenariju, za 20 ili 30 godina od sada, želeo bih — s obzirom na bilo kakvu superbakturu — da budem u mogućnosti da brzo proizvedem najbolje nanočestice koje mogu da je leče.
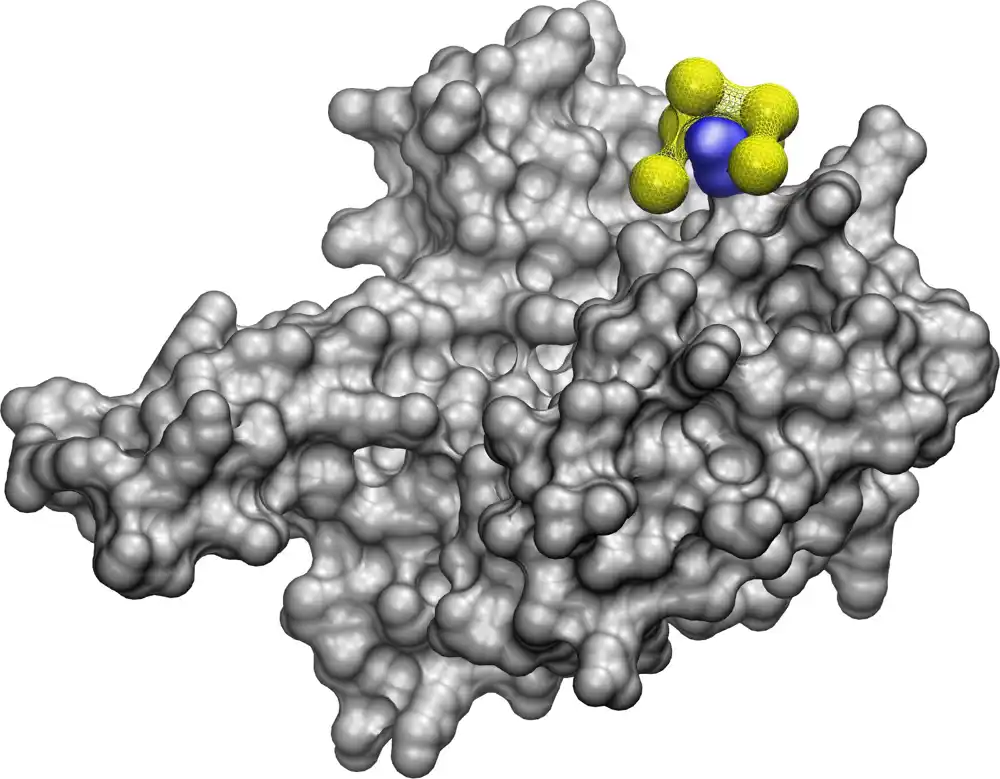
Veliki deo posla unutar ćelija obavljaju proteini. Mesta interakcije na njihovim površinama mogu spojiti molekule zajedno, razdvojiti ih i izvršiti druge modifikacije – otvaranje vrata u ćelije, razbijanje šećera da bi se oslobodila energija, građenje struktura koje podržavaju grupe ćelija i još mnogo toga. Ako bismo mogli da dizajniramo lekove koji ciljaju na ključne proteine u bakterijama i virusima, a da ne oštetimo naše ćelije, to bi omogućilo ljudima da se brzo bore protiv novih i promenljivih bolesti.
Novi model, nazvan NeCLAS, koristi mašinsko učenje — AI tehniku koja pokreće virtuelnog pomoćnika na vašem pametnom telefonu i ChatGPT-u. Ali umesto da nauči da obrađuje jezik, on apsorbuje strukturne modele proteina i njihova poznata mesta interakcije. Iz ovih informacija uči da ekstrapolira kako proteini i nanočestice mogu da interaguju, da predvidi mesta vezivanja i verovatnoću vezivanja između njih — kao i da predvidi interakcije između dva proteina ili dve nanočestice. Zasluge: Univerzitet u Mičigenu
„Postoje i drugi modeli, ali naš je najbolji za predviđanje interakcija između proteina i nanočestica“, rekao je Paolo Elvati, naučni saradnik UM u mašinstvu.
AlphaFold, na primer, je široko korišćen alat za predviđanje 3D strukture proteina na osnovu njegovih gradivnih blokova, nazvanih aminokiseline. Iako je ovaj kapacitet ključan, ovo je samo početak: otkrivanje kako se ovi proteini sklapaju u veće strukture i dizajniranje praktičnih sistema nanorazmera su sledeći koraci.
„Tu dolazi NeCLAS“, rekao je Jacob Saldinger, student doktorskih studija hemijskog inženjerstva na U-M i prvi autor studije. „Ona ide dalje od AlphaFold-a tako što pokazuje kako će nanostrukture međusobno komunicirati, i nije ograničeno na proteine. Ovo omogućava istraživačima da razumeju potencijalne primene nanočestica i optimizuju njihov dizajn.“
Tim je testirao tri studije slučaja za koje je imao dodatne podatke:
Dok mnogi proteinsko-proteinski modeli postavljaju aminokiseline kao najmanju jedinicu koju model mora uzeti u obzir, ovo ne funkcioniše za nanočestice. Umesto toga, tim je postavio veličinu te najmanje osobine da bude otprilike veličina aminokiseline, ali je onda pustio kompjuterski model da odluči gde su bile granice između ovih minimalnih karakteristika. Rezultat su reprezentacije proteina i nanočestica koje pomalo liče na kolekcije međusobno povezanih perli, pružajući veću fleksibilnost u istraživanju interakcija malih razmera.
„Pored toga što je opštiji, NeCLAS takođe koristi mnogo manje podataka o obuci od AlphaFold-a. Imamo samo 21 nanočesticu da pogledamo, tako da moramo da koristimo podatke o proteinima na pametan način,“ rekao je Matt Raimond, student doktorskih studija u oblasti elektrotehnike i računarstva. i koautor studije.
Zatim, tim namerava da istraži druge biofilmove i mikroorganizme, uključujući viruse.