U novoj studiji, istraživači sa IBM-a, Oksfordskog univerziteta i Diamond Light Source-a pokazuju da IBM-ov AI model, MoLFormer, može da generiše antivirusne molekule za višestruke ciljne virusne proteine, uključujući SARS-CoV-2, koji mogu ubrzati proces otkrivanja lekova i podstaći naše odgovor na buduće pandemije.
Rezultati su izloženi u novom radu objavljenom u Science Advances, a u vreme podnošenja rada, antivirusna svojstva jedanaest molekula uspešno su potvrdili istraživači sa Oksforda. Ovo otkriće ima potencijal da brže dovede lekove do ljudi u sledećoj krizi i omogući lečenje hitnih, životno opasnih bolesti na dohvat ruke.
Na početku pandemije, grupa kompjuterskih naučnika u IBM-u želela je da istraži da li bi generativna veštačka inteligencija mogla da se koristi za dizajniranje nikad viđenih molekula za blokiranje SARS-CoV-2, virusa koji izaziva COVID-19. Dejvid Stjuart, šef odseka za strukturnu biologiju na Odeljenju za kliničku medicinu na Univerzitetu u Oksfordu i direktor prirodnih nauka u Diamond Light Source, nacionalnom sinhrotronu u Velikoj Britaniji koji je autoritet za patogene HIV, SARS i ebolu, između ostalih virusa objašnjava da je u početku bio skeptičan. „Ideja da biste mogli da uzmete sekvencu proteina i, pomoću veštačke inteligencije, izvadite iz vazduha hemikalije koje bi se vezale za 3D mesto na virusu, izgledala je malo verovatna“, rekao je on.
Međutim, on i Martin Valsh, takođe stručni strukturni biolog i zamenik direktora za životne nauke u Diamondu, pridružili su se timu IBM-a i tokom tri godine pokazali da generativna veštačka inteligencija može da „izvuče održive početne tačke za antivirusne lekove iz ničega, “ u saradnji sa Enamine Ltd., dobavljačem hemikalija u Ukrajini, i drugim istraživačima sa Oksforda.
Pošto je generativni model bio i osnovni model, prethodno obučen na ogromnim količinama sirovih podataka, bio je dovoljno svestran da stvori nove inhibitore za više proteinskih ciljeva bez dodatne obuke ili bilo kakvog znanja o njegovoj 3D strukturi.
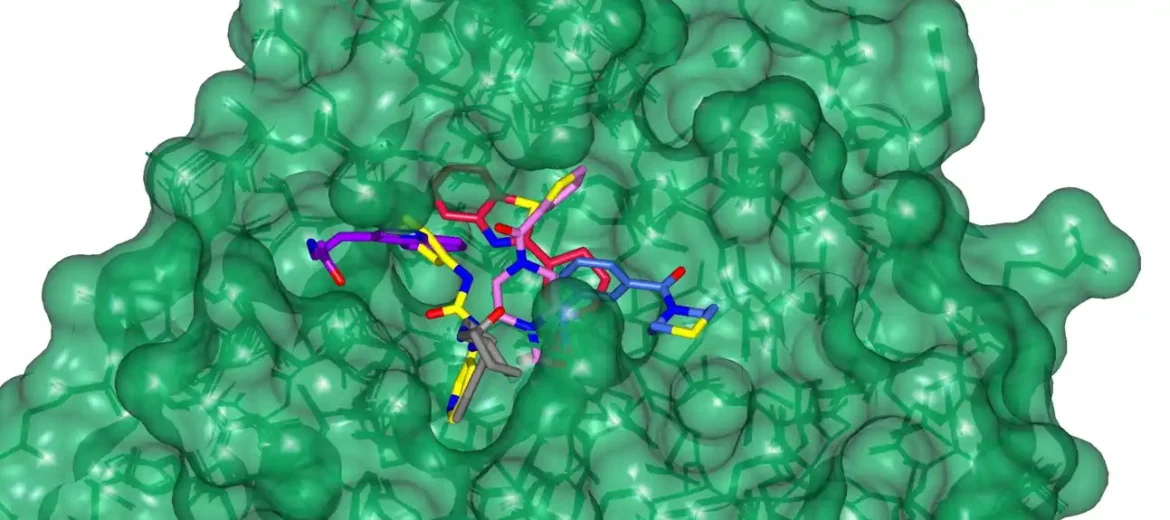
Grupe Stjuarta i Volša počele su da rade na dva esencijalna proteina SARS-CoV-2, a to su spike protein i glavna proteaza. Koristeći ove mete, tim je pogodio četiri potencijalna antivirusna leka COVID-19 za delić vremena koje bi bilo potrebno konvencionalnim metodama. Rad je zatim iskoristio Diamondove visoko propusne makromolekularne kristalografske linije da bi se vizuelizovao kako podskup AI generiše jedinjenja vezana za glavnu proteazu.
Njihov rad je prikazan u njihovom novom dokumentu u Science Advances, a IBM je izdao interfejs zasnovan na vebu za interakciju sa modelima i modelima hemijske osnove poput IBM Cloud-a.
Tim je naveo da validirani molekuli imaju mnogo više prepreka za uklanjanje, uključujući klinička ispitivanja, pre nego što ih kompanije potencijalno pretvore u lekove. Ali čak i ako se „pogoci“ generisani veštačkom inteligencijom nikada ne materijalizuju u stvarne lekove, rad pruža potvrdu da generativna AI ima važnu ulogu u budućnosti razvoja lekova, posebno u vreme krize.
„Trebalo je vremena da se razviju i validiraju ove metode, ali sada kada imamo radni cevovod, možemo da generišemo rezultate mnogo brže“, rekao je ko-autor studije, Pajel Das, istraživač u IBM Research-u. „Kada se pojavi sledeći virus, generativna AI bi mogla biti ključna u potrazi za novim tretmanima.
„Generisanje početnih jedinjenja koja se sa visokim afinitetom vezuju za metu leka od interesa ubrzava proces otkrivanja lekova zasnovanog na strukturi i podupire naše napore da budemo bolje pripremljeni za buduće pandemije“, rekao je Martin Volš, koji je bio ko-stariji autor u Diamondu.
Istraživači su izgradili svoj model, kontrolisano stvaranje molekula (ili CogMol), na generativnoj arhitekturi veštačke inteligencije poznatoj kao varijacioni autoenkoderi ili VAE. VAE kodiraju sirove podatke u komprimovani prikaz, a zatim ih dekodiraju ili prevode nazad u statističku varijaciju originalnog uzorka. Njihov model je obučen na velikom skupu podataka molekula predstavljenih kao nizovi teksta, zajedno sa opštim informacijama o proteinima i njihovim osobinama vezivanja. Ali namerno su izostavili informacije o 3D strukturi SARS-CoV-2 ili molekulima za koje se zna da se vezuju za nju. Njihov cilj je bio da svom generativnom modelu temelja daju široku bazu znanja kako bi se mogao lakše primeniti za zadatke molekularnog dizajna koje nikada ranije nisu videli.
Njihov cilj je bio da pronađu molekule slične leku koji bi se vezali za dve mete COVID proteina: šiljak, koji prenosi virus do ćelije domaćina, i glavnu proteazu, koja pomaže u njegovom širenju. Iako su do tada otkrivene 3D strukture oba proteina, istraživači IBM-a su odlučili da koriste samo njihove aminokiselinske sekvence, izvedene iz njihove DNK. Ograničavajući se na ovaj način, nadali su se da bi model mogao naučiti da generiše molekule bez poznavanja oblika svoje mete.
Istraživači unose samo sekvencu aminokiselina za svaki cilj proteina u CogMol, koji je generisao 875.000 molekula kandidata za tri dana. Da bi suzili opseg, istraživači su proveli kandidate kroz platformu za retrosintezu, IBM RKSN for Chemistri, da bi razumeli koji će sastojci biti potrebni za sintezu jedinjenja. Na osnovu predviđenih recepata platforme, odabrali su 100 molekula za svaku metu. Hemičari u Enaminu su dalje uparili listu na četiri molekula za svaku metu, birajući one za koje se smatra da su najlakše za proizvodnju.
Nakon što je sintetizovao osam novih molekula, Enamine ih je poslao u Oksford radi testiranja njihove sposobnosti da poremete funkcije dve proteinske mete u laboratorijama profesora Krisa Šofilda i profesora Gevina Skritona. . Intenzivni rendgenski snop generisan iz dijamanta koji je 10 milijardi puta svetliji od sunca korišćen je da se vizuelizuje kako su jedinjenja u interakciji sa proteinima da bi deaktivirali njihovu funkciju. Nova jedinjenja su dalje testirana u testovima ciljne inhibicije i neutralizacije živih virusa. Dva validirana antivirusna leka ciljaju na glavnu proteazu; druga dva ne samo da su ciljala na spike protein, već su se pokazala sposobnim da neutrališu svih šest glavnih varijanti COVID-a. „Dobijate mapu koja tačno pokazuje gde se stvari vezuju, i prasak! Dobili ste potvrdu“, rekao je Stjuart.
CogMol je jedan od nekoliko modela hemijskih osnova koje je IBM od tada razvio. Najveći, MoLFormer-KSL, obučen je za bazu podataka od više od 1,1 milijardu molekula i trenutno ga koristi Moderna za dizajniranje mRNA lekova. „Stvorili smo valjane početne tačke za ubrzani razvoj antivirusnih lekova koristeći generativni temeljni model koji je znao relativno malo o svojim proteinskim ciljevima“, rekao je ko-stariji autor studije, Džejson Krejn, istraživač u IBM Research-u i profesor na Oksfordu. „Nadam se da će nam ove metode omogućiti da stvaramo antivirusna i druga hitno potrebna jedinjenja mnogo brže i jeftinije u budućnosti.“
Iako su se istraživači fokusirali na validaciju antivirusnih lekova za COVID, oni tvrde da se ove metode mogu proširiti na postojeće viruse koji nastavljaju da mutiraju, poput gripa, ili virusa koji tek treba da se pojave. „Ako želite da budete spremni za sledeću pandemiju, želite lekove koji deluju na različitim mestima proteina“, zaključio je Stjuart. „Virusu postaje mnogo teže da pobegne.“