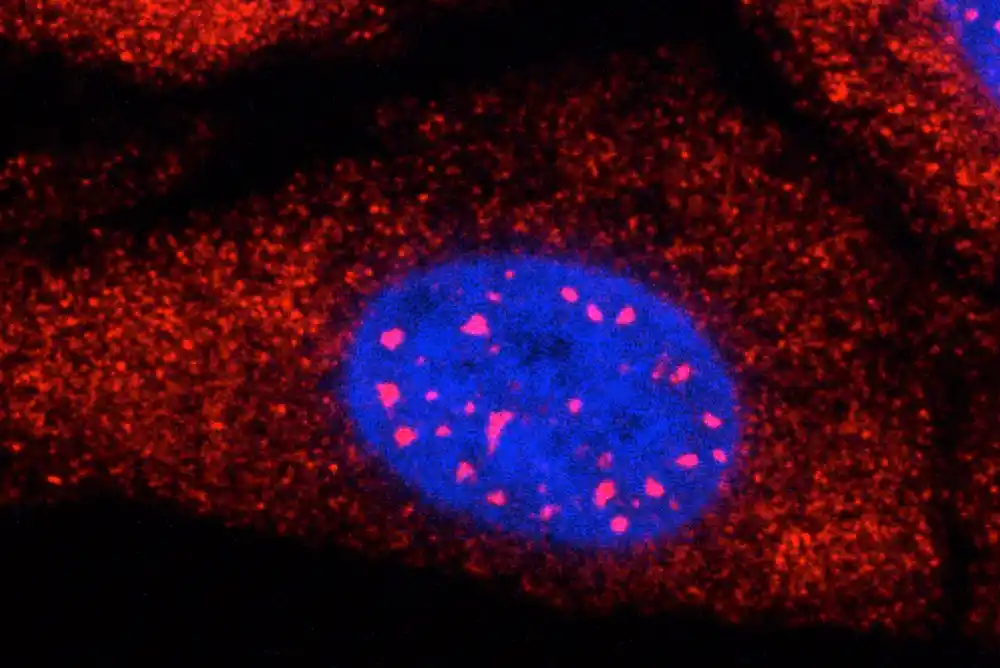
DNK, koja se često smatra „nacrtom života“, sadrži uputstva za izgradnju proteina koji su ćelijama potrebni da bi preživele i pravilno funkcionisale. Ali DNK nije savršena i može doći do grešaka tokom replikacije. Ponekad to može dovesti do toga da se isečci građevinskih blokova DNK koji se nazivaju nukleotidi — G (gvanin), A (adenin), T (timin), C (citozin) — ponavljaju previše puta zaredom.
Ovo može dovesti do vrste mutacije, poznate kao ekspanzije ponavljanja nukleotida, koja može da promeni funkciju i strukturu vitalnih proteina i može dovesti do retkih neurodegenerativnih stanja kao što su Hantingtonova bolest i amiotrofična lateralna skleroza (ALS).
Novo istraživanje člana Instituta Vajthed Ankur Jain, diplomirani student Rejčel Anderson i njihove kolege bliže razmatraju kako sekvenca ponavljanja uključena u Hantingtonovu bolest – ponavljanje CAG – dovodi do proizvodnje abnormalnih proteina koji se pogrešno sklapaju i nakupljaju unutar ćelija, začepljujući pokreće važne ćelijske procese.
Njihovi nalazi, objavljeni u časopisu Molecular Cell 30. januara, otkrivaju da prošireno ponavljanje CAG-a može ometati spajanje. Kao što je prikazano na ilustraciji ispod, ovo je proces gde se delovi RNK koji ne kodiraju proteine, poznati i kao introni, izrezuju. Preostale sekcije, zvane egzoni, se zatim spajaju kako bi se formirala konačna glasnička RNK koja nosi uputstva za izgradnju proteina.
Prema istraživačima, prošireno ponavljanje CAG-a stvara nove markere, ili mesta prihvatanja spajanja, što dovodi do sečenja i lepljenja genetskih informacija koje se dešavaju na različitim spojevima nego obično.
„Pitanje zašto mozgovi pacijenata sa poremećajima ponovljene ekspanzije imaju lažne proteine zbunilo je naučnike neko vreme“, kaže Džejn, koji je takođe docent biologije i profesor razvoja karijere Tomasa D. i Virdžinije V. Kabot na Univerzitetu. Massachusetts Institute of Technologi. „Sada, pošto imamo razumevanje molekularnog mehanizma, možemo pokušati da ciljamo put spajanja i smanjimo proizvodnju ovih proteina.“
Rasklapanje RNK ukosnica
RNK je manje stabilna od DNK, a uobičajeni pristupi analizi RNK se oslanjaju na enzim koji se zove reverzna transkriptaza. Iako se obično u ćeliji DNK čita u RNK, ovaj enzim čita RNK molekule u komplementarni lanac DNK (cDNK). Ovo omogućava istraživačima da pažljivo analiziraju sekvence RNK bez rizika od degradacije genetskih informacija.
Ali reverzna transkripcija RNK koja sadrži ponavljanje dolazi sa sopstvenim izazovima — ovi molekuli imaju tendenciju da se savijaju, formirajući petlje za ukosnicu, a kada se ove petlje ne odmotaju u potpunosti tokom reverzne transkripcije, istraživači ostaju sa prazninama i greškama u cDNK.
U novom radu, Jain i Anderson su koristili drugačiji pristup za osetljivu reverznu transkripciju RNK koje sadrže ponavljanje u cDNK. Konkretno, istraživači su radili sa enzimom zvanim TGIRT (termostabilna intronska reverzna transkriptaza II grupe) koji ostaje aktivan na visokim temperaturama, omogućavajući mu da razbije strukture ukosnica i uhvati sekvence koje sadrže ponavljanje sa većom vernošću.
„Kada zagrejete jaje, ono postaje žuto jer se proteini u jajetu razvijaju zbog visoke temperature. Mi koristimo istu stvar, ali sa RNK strukturama“, kaže Anderson.
Zatim su istraživači počeli da mapiraju ova ponavljanja na referentni genom, koji služi kao vodič za genetske informacije kod čoveka, ali su brzo naišli na izazove. „Slova“ koja čine ljudski genom G-A-T-C kombinuju se u različitim sekvencama da bi formirali lance DNK u našim ćelijama.
To znači da su ponovljeni obrasci u ljudskom genomu neizbežni (bolesti zasnovane na ponavljanju nastaju samo kada se jedna sekvenca — kao što je CAG — ponavlja previše puta zaredom) i svaki obrazac se može pojaviti na više lokacija u genomu. Dakle, određivanje gde je nastala RNK koja sadrži ponavljanje je kao rekonstruisanje priče iz fragmentiranih rečenica bez konteksta.
„Tada smo odlučili da drugačije pristupimo mapiranju ponavljanja“, kaže Anderson. Istraživači su razvili novi alat, nazvan SATCfinder, koji bira RNK sekvence sa najmanje tri CAG ponavljanja. Ova ponavljanja se zatim kompjuterski skraćuju, a ostatak sekvence se mapira na referentni genom. Lokacija, ili koordinate mapiranja, uzorka neposredno pre praćenja CAG ponavljanja, omogućavajući istraživačima da shvate tačno kuda bi ponavljanja trebalo da idu.
Pažljivije razmatranje spajanja
Prethodni rad iz laboratorije Jain je pokazao da kada RNK koje sadrže ponavljanje napuste jezgro i stignu u ćelijsku citoplazmu, formiraju grudve slične gelu.
Tipično, u citoplazmi, RNK stupaju u interakciju sa ćelijskom mašinerijom koja traži marker na RNK, koji se naziva početni kodon, da bi započeo prevođenje instrukcija za izgradnju proteina. Istraživači su teoretizirali da bi RNK koje sadrže ponavljanje mogle zbuniti mašineriju, utičući na nju da prevodi uputstva sa različitih polaznih tačaka. Ovaj proces, nazvan RAN translacija, tada bi mogao dovesti do stvaranja nepotrebnih proteina koji ne samo da su skloni zgrušavanju, već i doprinose gumiranju RNK u citoplazmi.
Ali ovo objašnjenje nije bilo sasvim zadovoljavajuće za Jaina i Andersona, i oni su želeli da saznaju više o tome zašto RNK koje sadrže ponavljanje uopšte dovode do slučajnog prevoda instrukcija. Da bi to istražili, napravili su skup sekvenci sa šablonom „CAG“ ponovljenim 240 puta uzastopno. Baš kao što su očekivali, kada su ove sekvence dospele u citoplazmu, počele su da se agregiraju.
Kada su istraživači izvršili sekvenciranje RNK na ovim ćelijama i analizirali rezultate pomoću SATCfinder-a, pronašli su svoj odgovor: CAG ponavljanja u RNK su često bila spojena u neočekivane sekvence, dalje od ponavljanja u DNK, sa isečenim regionima. To je značilo da je prisustvo CAG ponavljanja više puta zaredom dovelo do stvaranja novih mesta za sečenje i lepljenje na ivicama samog ponavljanja, stvarajući abnormalne RNK transkripte koji zatim proizvode proteine koji se pogrešno savijaju i spajaju zajedno.
Sada su istraživači u Jain laboratoriji zainteresovani da dalje istraže kako prošireno ponavljanje CAG-a izaziva greške u spajanju. Takođe se nadaju da će saznati više o tome u kojoj meri ove greške spajanja doprinose patologiji stanja kao što je Hantingtonova bolest.
„Postoji niz mehanizama koji se spajaju i doprinose ćelijskoj smrti u Hantingtonovoj. Ovo je jedan deo slagalice koji doprinosi našem molekularnom razumevanju kako ove ponavljanja iskrivljuju ćelijske funkcije“, kaže Džejn.