Uprkos sve većoj upotrebi genomskog sekvenciranja u kliničkoj praksi, tumačenje retkih genetskih mutacija, čak i među dobro proučenim genima bolesti, ostaje teško. Trenutni prediktivni modeli su korisni za tumačenje tih mutacija, ali su skloni da pogrešno klasifikuju one koje ne izazivaju bolesti, što doprinosi lažno pozitivnim rezultatima.
Istraživači sa Maks Plank instituta za molekularnu ćelijsku biologiju i genetiku (MPI-CBG) u Drezdenu, Centra za sistemsku biologiju Drezden (CSBD) u Nemačkoj i Harvardske medicinske škole u Bostonu, SAD, razvili su alat pod nazivom Dešifrovanje mutacija u Actionable Genes (DeMAG). Istraživanje je objavljeno u časopisu Nature Communications.
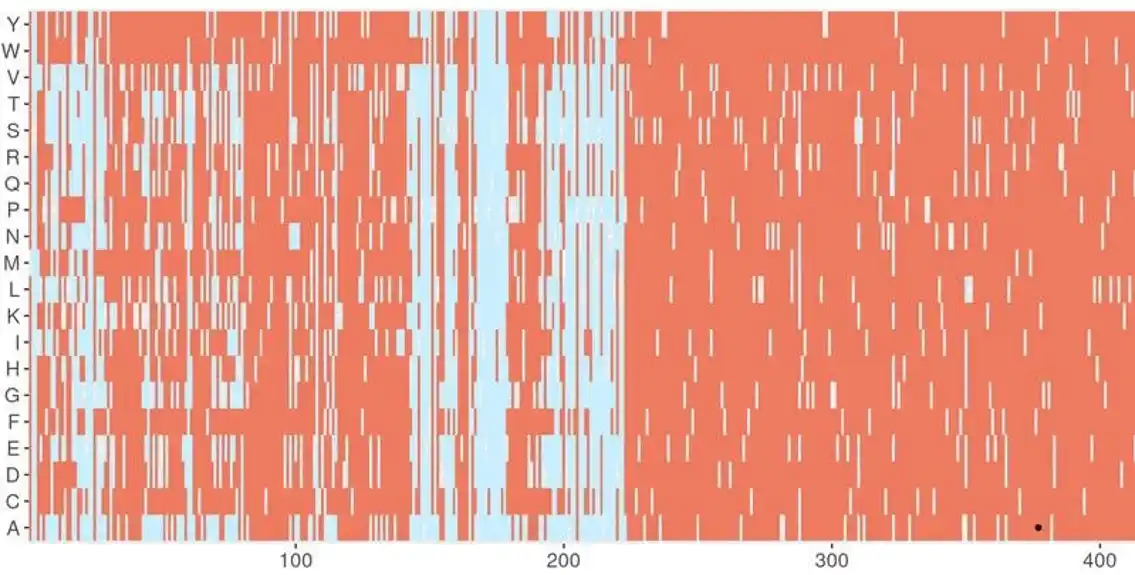
DeMAG je veb server otvorenog koda (demag.org) koji nudi tumačenje efekata svih potencijalnih mutacija pojedinačnih aminokiselina koje bi se mogle javiti u 316 klinički relevantnih gena koji uzrokuju bolesti za koje su već dostupne preventivna dijagnostika i tretmani. DeMAG pruža medicinskim stručnjacima alat koji im omogućava da preciznije procene efekat mutacija u tim genima smanjujući stopu lažno pozitivnih, što znači da se manje benigne mutacije predviđaju kao patogene. Kao rezultat toga, alat može podržati kliničko donošenje odluka.
Poslednjih godina, genomsko sekvenciranje je postalo jeftinije i naprednije. S jedne strane, ovo omogućava kliničarima da sve više koriste sekvenciranje u dijagnostičke svrhe, a istovremeno omogućava naučnicima da istraže više hipoteza istraživanja. S druge strane, mnoge otkrivene mutacije nemaju jasnu kliničku interpretaciju. Neizvesnost oko toga da li mutacija izaziva bolest može biti stresna za pacijente i dovesti do psihološkog opterećenja, morbiditeta i troškova zdravstvene nege koji su povezani sa nedovoljnom i preteranom dijagnozom.
Iako se postojeći alati već koriste za predviđanje funkcionalnog uticaja ovih varijanti, njihov učinak je pristrasan zbog ograničenih kliničkih podataka koji otežavaju razlikovanje patogenih (koje izazivaju bolest) i benignih (neutralnih) varijanti unutar datog gena i često dovode do pogrešne klasifikacije. mutacije koje ne izazivaju bolest kao patogene. Rješavanje ovih poteškoća je ključno za razvoj pouzdanog prediktora za kliničke primjene.
Istraživačka grupa Agnes Toth-Petroczi na MPI-CBG i CSBD udružila se sa Kristoferom Kasom, docentom medicine na Odeljenju za genetiku Brigama i ženske bolnice na Medicinskom fakultetu Harvarda, i Ivanom Adžubejem, naučnim saradnikom na Odseku biomedicinske informatike na Harvard Medical School, da razvije statistički model i veb server DeMAG koji postiže visoku tačnost u interpretaciji genetskih mutacija u genima bolesti. Da bi to uradili, istraživači su pažljivo odabrali poznate patogene i benigne mutacije za obuku modela.
„Koristili smo kliničke i različite baze podataka o populaciji. Odabrali smo samo mutacije čije je kliničko tumačenje dogovoreno među više podnosilaca kao što su lekari i genetičke laboratorije. Takođe smo uključili podatke o predacima koji su nedovoljno zastupljeni u trenutnim bazama podataka o populaciji, kao što su korejski ili japanski, da bude još reprezentativniji i tačniji“, objašnjava Federika Lupino, prvi autor istraživačkog rada i dr. student u grupi Tot-Petroči.
DeMAG uključuje novu karakteristiku, „skor partnera“, koja identifikuje grupe aminokiselina u proteinu koje dele isti klinički efekat. Sa rezultatom partnera, DeMAG koristi prednosti odnosa aminokiselina zasnovanih na evolucionim informacijama iz genoma mnogih organizama i nedavnoj AI (veštačkoj inteligenciji) revoluciji predviđanja 3D oblika proteina koristeći algoritam AlphaFold koji je razvio Google DeepMind.
Agnes Toth-Petroczi, koja je nadgledala studiju, zaključuje: „Pružamo osnovni okvir za integraciju kliničkih i proteinskih podataka kako bismo pomogli u proceni uticaja mutacija. Nadamo se da će naš alat i veb server olakšati procenu efekata varijanti i donošenje kliničkih odluka Štaviše, novorazvijene karakteristike se mogu primeniti na druge gene i organizme izvan ljudi.“ DeMAG kod je dostupan na GitLab-u i svi podaci su slobodno dostupni na veb serveru na demag.org.