Viruse je teško pratiti. Oni brzo evoluiraju i redovno razvijaju nove proteine koji im pomažu da zaraze svoje domaćine. Ove brze promene znače da istraživači i dalje pokušavaju da shvate mnoštvo virusnih proteina i precizno kako oni povećavaju sposobnost virusa da inficiraju – znanje koje bi moglo biti ključno za razvoj novih ili boljih tretmana za borbu protiv virusa.
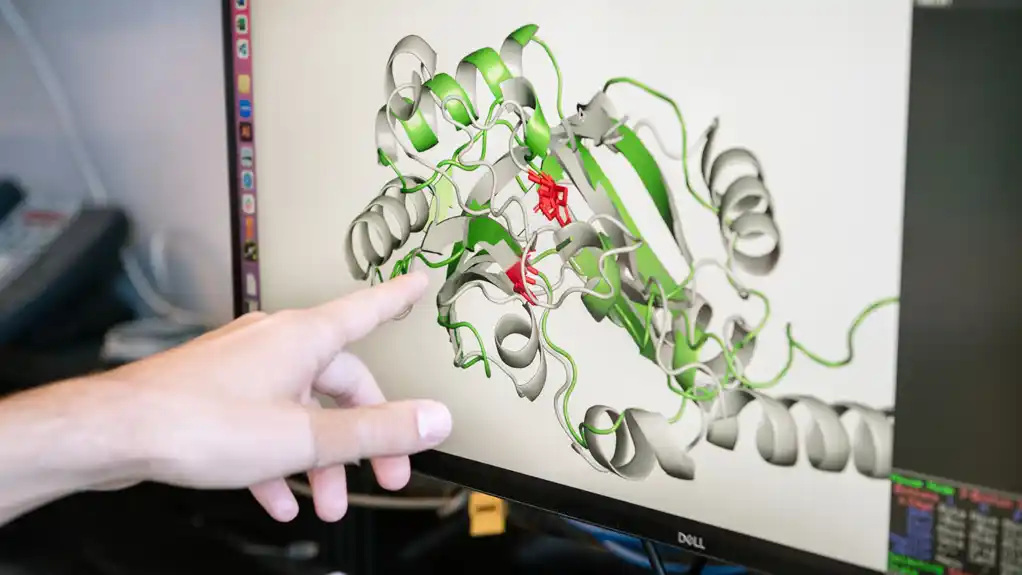
Sada je tim naučnika sa Instituta Gledston i Instituta za inovativnu genomiku, predvođen dr Dženifer Doudna, iskoristio računarske alate da predvide trodimenzionalne oblike skoro 70.000 virusnih proteina.
Istraživači su uparili 3D oblike sa strukturama proteina čije su funkcije već poznate. Pošto struktura proteina direktno doprinosi njegovoj biološkoj funkciji, njihova studija pruža nove uvide u to šta tačno rade ovi manje poznati proteini.
Među njihovim drugim nalazima, objavljenim u časopisu Priroda, istraživači su otkrili moćan način na koji virusi izbegavaju imuni sistem. U stvari, otkrili su da virusi koji inficiraju bakterije i oni koji inficiraju više organizme — uključujući ljude — dele sličan, drevni mehanizam za izbegavanje imunološke odbrane domaćina.
„Kako se pojavljuju virusi sa pandemijskim potencijalom, važno je utvrditi kako će stupiti u interakciju sa ljudskim ćelijama“, kaže Doudna, koja je takođe profesor na UC Berkliju i istraživač na Medicinskom institutu Hauarda Hjuza. „Naša nova studija pruža alat za predviđanje šta ti novonastali virusi mogu da urade.
Obično, da bi otkrili funkciju proteina, istraživači će tražiti sličnosti između njegove različite sekvence aminokiselinskih „građevinskih blokova“ i sekvenci aminokiselina drugih proteina sa poznatim funkcijama. Međutim, pošto virusi evoluiraju tako brzo, mnogim virusnim proteinima nedostaju jake sličnosti sa poznatim proteinima.
Ipak, baš kao što se različite kombinacije građevinskih blokova mogu koristiti za konstruisanje veoma sličnih struktura, proteini sa različitim sekvencama mogu deliti 3D oblike i igrati slične biološke uloge.
„Razmatrali smo sličnosti između oblika proteina kao obećavajuću alternativu za određivanje funkcije virusnih proteina“, kaže dr Džejson Nomburg, postdoktorski naučnik u Doudninoj laboratoriji u Gledstonu i prvi autor studije. „Pitali smo: Šta možemo naučiti iz proteinskih struktura koje bismo mogli propustiti kada samo razmatramo sekvence?“
Da bi odgovorio na to pitanje, tim se okrenuo istraživačkoj platformi otvorenog pristupa pod nazivom AlphaFold, koja predviđa 3D oblik proteina na osnovu njegove sekvence aminokiselina. Koristili su AlphaFold da predvide oblike 67.715 proteina iz skoro 4.500 vrsta virusa koji inficiraju eukariote (organizme uključujući biljke, životinje i ljude koji sadrže DNK u jezgru svojih ćelija). Zatim su, koristeći alat za duboko učenje, uporedili predviđene strukture sa onima poznatih proteina drugih virusa, kao i nevirusnih proteina iz eukariota.
„Ovo ne bi bilo moguće bez nedavnog napretka u ovim vrstama računarskih alata koji nam omogućavaju da precizno i brzo predvidimo i uporedimo strukture proteina“, kaže Nomburg.
Tim je otkrio da 38 procenata novopredviđenih oblika proteina odgovara prethodno poznatim proteinima i da je pronašao ključne veze između njih.
Na primer, neke od novopredviđenih struktura pripadaju grupi takozvanih „proteina sličnih UL43“, koji se nalaze u humanim herpes virusima, uključujući one koji izazivaju mononukleozu i vodene boginje.
„Ovi novi virusni proteini izgledaju šokantno slični poznatim nevirusnim proteinima u ćelijama sisara koji pomažu u transportu građevinskih blokova DNK i RNK kroz membrane“, kaže Nomburg. „Pre ovog rada, nismo znali da ovi proteini mogu funkcionisati kao transporteri.“
Tim je takođe pronašao podudaranja između novopredviđenih struktura virusnih proteina i struktura drugih virusnih proteina. Najvažnije, analiza je otkrila strategiju za izbegavanje imunološke odbrane domaćina koja se široko deli na viruse koji inficiraju životinje i viruse poznate kao fagi koji inficiraju bakterije. Čini se da je ovaj mehanizam sačuvan tokom evolucije.
„Ovo ulazi u veoma uzbudljivo područje jer postoji sve više dokaza da urođeni imunitet složenih organizama, uključujući ljude, podseća na mnoge različite vrste urođenog imuniteta kod bakterija“, kaže Nomburg. „Mi ćemo dublje istražiti ove evolutivne veze, jer bolje razumevanje načina na koji naše ćelije reaguju na viruse može dovesti do novih pristupa za poboljšanje antivirusne odbrane.
U međuvremenu, tim je učinio javno dostupnim 70.000 novopredviđenih struktura virusnih proteina, kao i podatke iz njihovih novih analiza. Ovi resursi bi mogli pružiti drugim istraživačima priliku da otkriju dodatne strukturne veze između proteina koje produbljuju znanje o tome kako virusi komuniciraju sa svojim domaćinima.
„Iz perspektive borbe protiv bolesti, ovaj rad je uzbudljiv jer ističe nove moguće pristupe za dizajniranje široko efikasnih antivirusnih terapija“, kaže Doudna. „Na primer, pronalaženje uobičajenih, očuvanih načina na koje virusi izbegavaju imunitet može dovesti do moćnih antivirusnih lekova koji su efikasni protiv mnogo različitih virusa odjednom.“